大黄鱼(Larimichthys,crocea)ISG15基因单核苷酸多态性及与抗病性状的相关性研究*
李凤欣 殷小龙 卢德政 刘 成 张建设 沈 斌
大黄鱼()基因单核苷酸多态性及与抗病性状的相关性研究*
李凤欣1殷小龙2卢德政1刘 成1张建设1沈 斌1①
(1. 浙江海洋大学 国家海洋设施养殖工程技术研究中心 浙江舟山 316022; 2. 浙江省舟山市水产研究所 浙江舟山 316111)
由哈维氏弧菌()等细菌感染引起的弧菌病对我国大黄鱼()的养殖造成了严重危害。通过哈维氏弧菌人工感染大黄鱼建立易感组和抗病组, 采用PCR扩增和直接测序法对大黄鱼干扰素刺激基因双拷贝(和)进行单核苷酸多态性(SNPs)检测和分型, 并与其哈维氏弧菌抗性进行关联分析。结果表明, 从大黄鱼和基因中分别筛选到10个和4个SNP位点并进行了成功分型。经统计分析,基因的186 G/C和318 C/T位点以及基因的297 G/T位点的基因型频率和等位基因频率在易感群体和抗病群体中均存在极显著差异, 表明这3个SNP位点与大黄鱼哈维氏弧菌抗性显著相关。连锁不平衡分析结果显示,的SNPs可形成1个单倍块和11种单倍型, 而的SNPs可形成1个单倍块和5种单倍型。其中,基因的单倍型H2(CCCCGGTACC)、H6(TCCCACTGTC)和H9(TCCCAGTGCC)与大黄鱼哈维氏弧菌抗性显著相关;基因的单倍型H1(CCCG)和H4(TCCG)与大黄鱼哈维氏弧菌抗性极显著相关。这些和基因的SNP位点以及单倍型可以作为抗哈维氏弧菌病大黄鱼选育的候选分子标记。
大黄鱼(); 干扰素刺激基因; 单核苷酸多态性; 哈维氏弧菌
由干扰素(Interferons, IFNs)介导的天然免疫在生物体抗病毒感染方面发挥重要的作用。当干扰素与细胞表面的受体结合后, 可以通过JAK-STAT信号通路引发级联信号放大反应, 诱导数以百计的干扰素刺激基因(Interferon-stimulated genes, ISGs)表达, 进而发挥抗病毒免疫功能(Schneider, 2014)。在众多已报道的干扰素刺激基因中, 干扰素刺激基因15 (Interferon-stimulated gene 15,)是最早被报道的干扰素刺激基因(Blomstrom, 1986)。ISG15蛋白包含3个结构域: 两个串联的泛素样结构域(ubiquitin-like domain)、短连接序列(short linker)和羧基端LRGG基序(LRGG conjugation motif) (Zhang, 2011)。胞内ISG15前体蛋白能够通过泛素激活酶(E1)-泛素结合酶(E2)-泛素连接酶(E3)三酶级联反应共价结合到靶蛋白上并对靶蛋白进行ISG化修饰(ISGylation) (嵇祝星等, 2022), 进而发挥抗病毒作用。另外, ISG15单体蛋白还可以作为细胞因子被淋巴细胞、单核细胞等分泌到胞外, 发挥诱导淋巴细胞增殖和T淋巴细胞产生干扰素等功能(D’Cunha, 1996)。
硬骨鱼类同样具有基因。O’Farrell等(2002)首先从虹鳟()的白细胞cDNA文库中鉴定到了鱼类的基因。随后, 学者们相继对鲫鱼() (Liu, 2002)、斜带石斑鱼() (Huang, 2013)、草鱼() (Dai, 2017)等10多种鱼类的基因开展了研究。鱼类的ISG15同样包含3个结构域, 即两个串联的泛素样结构域、短连接序列和LRGG基序(Shen, 2019)。研究结果表明, 病毒和细菌感染(Wang, 2012)均能够有效诱导鱼类的表达量上调。鱼类的胞内ISG15蛋白也能与靶蛋白共价结合进行ISG化修饰(Liu, 2002)。鱼类ISG15单体蛋白也能够作为细胞因子被头肾淋巴细胞分泌到胞外(Wang, 2012)。这些结果有力地证明鱼类在抗病毒和细菌天然免疫方面发挥重要的作用。
大黄鱼()隶属于鲈形目(Perciformes)、石首鱼科(Sciaenidae), 是我国东部沿海地区最重要的经济养殖鱼类之一(Liu, 2008)。随着大黄鱼养殖规模的迅速扩大以及集约化程度的不断提高, 各种养殖疾病不断出现且日趋严重(唐嘉嘉等, 2022)。在细菌性疾病中, 由哈维氏弧菌()等弧菌属细菌感染引起的弧菌病是大黄鱼网箱养殖过程中最为常见的病害, 对大黄鱼的养殖造成严重的危害(徐晓津等, 2010)。随着分子生物学技术的不断发展, 越来越多的学者开始开展大黄鱼抗弧菌感染免疫分子机制方面的研究。近年来, 学者们先后对大黄鱼溶菌酶(Zheng, 2007)、铁调素(Hepcidin) (Wang, 2009)、Toll样受体(Huang, 2011)等免疫相关基因开展了研究, 探讨了这些基因在大黄鱼抗弧菌感染免疫方面的功能。关于基因方面, Shen等(2019)首次从大黄鱼中鉴定到了2个基因拷贝(和), 研究结果表明大黄鱼的2个基因拷贝均在抗病毒和细菌天然免疫方面发挥作用。
利用分子标记辅助选择培育高产优质抗逆良种, 已经成为大黄鱼育种的重要手段之一(王志勇, 2014)。单核苷酸多态性(single nucleotide polymorphisms, SNPs)作为一种新型的分子遗传标记, 被广泛应用于动植物的分子遗传选育(王晨阳等, 2019)。目前已经有相关学者在大黄鱼中开展了与生长(张玉等, 2018)、抗逆(陈小明等, 2017)、抗病(孙明洁等, 2019)等性状相关的SNP位点挖掘及关联分析工作。这些研究结果, 为大黄鱼优质抗逆新品种的遗传选育提供了理论依据。本研究在前期分子克隆和基因表达基础上, 进一步对大黄鱼和基因的SNP位点进行筛选, 开展和基因多态性与哈维氏弧菌抗性的相关性分析, 以期为大黄鱼抗病选育提供理论基础。
1.1 样品采集
试验所用大黄鱼采自浙江省舟山市水产研究所, 系该所用良种场保存的大黄鱼亲本繁育的用于增殖放流的F1代苗种。哈维氏弧菌菌株购自中国普通微生物菌种保藏管理中心, 鱼类致病菌。随机采集400尾大黄鱼(体重约25 g), 暂养于海水温度25 °C、盐度25、pH 8.5、持续充气的水族箱中。每天换水1次, 投饵2次, 适应1周确认大黄鱼健康后开始实验。大黄鱼抗病和易感群体获得的具体方法参考文献(柴欣等, 2017), 人工感染哈维氏弧菌的具体方法参考文献(Shen, 2019)。将哈维氏弧菌菌种接种于含有2% NaCl的LB培养基中, 28 °C培养至对数中期。将菌液进行离心, 用无菌PBS洗涤2次, 重悬制成菌悬液并调整浓度至1×107CFU/mL。取暂养1周后的健康大黄鱼400尾, 腹腔注射1×107CFU/mL浓度的菌悬液(剂量0.5 mL/100 g鱼体重)。注射1 d后试验鱼开始出现死亡个体, 注射3 d后死亡个体数不断上升, 至第8天死亡个体数达到最大, 随后死亡个体数开始逐渐下降并趋于稳定, 注射10 d以后无死亡个体出现。注射10 d内共死亡大黄鱼263尾, 死亡率达到65.75%。将哈维氏弧菌感染10 d内死亡并具有体表充血溃烂、肝脾肿大等明显病症的大黄鱼个体作为易感组, 感染10 d后仍然存活的个体作为抗病组。分别采集易感组(100尾)和抗病组(100尾)个体的肌肉组织放入无水乙醇中, 保存于–20 °C冰箱备用。
1.2 基因组DNA提取
利用天根生化科技(北京)有限公司的血液/细胞/组织基因组DNA提取试剂盒提取肌肉组织的基因组DNA。每尾个体取约30 mg肌肉组织, 根据试剂盒说明书步骤进行基因组DNA提取, 提取过程中注意避免基因组DNA污染。利用1%的琼脂糖凝胶电泳检测基因组DNA提取质量。提取好的基因组DNA样品保存于–20 °C冰箱备用。
1.3 ISG15-1和ISG15-2基因SNP位点的筛选
从GenBank数据库下载大黄鱼和基因的基因组序列(序列号: NC_040013), 选取和基因的5’和3’侧翼序列, 利用Primer premier 5软件设计2对PCR扩增引物(表1)。从易感组和抗病组中分别随机选择10尾大黄鱼个体, 以基因组DNA为模板, 对和基因进行PCR扩增。PCR反应体系为20 μL, 包括: 10 μL Premix TaqTMDNA聚合酶(TaKaRa), 0.4 μL上游引物(10 μmol/L), 0.4 μL下游引物(10 μmol/L), 0.4 μL基因组DNA (50 ng/μL)以及8.8 μL ddH2O。PCR反应条件: 95 °C预变性5 min, 32个扩增循环(95 °C变性30 s, 56 °C退火30 s, 72 °C延伸40 s), 最后72 °C延伸10 min。取少量PCR产物利用1%琼脂糖凝胶电泳进行PCR产物确认, 最后将PCR产物送测序公司进行测序。利用MEGA4软件对扩增产物的测序结果进行比对, 筛选出等位基因频率大于20%的位点作为候选SNP位点(柴欣等, 2017)。对筛选到的SNP位点进行统计, 并参考ISG15蛋白的功能结构域(Shen, 2019), 对SNP位点进行功能结构域定位分析。

表1 大黄鱼ISG15-1和ISG15-2基因多态性检测引物
1.4 ISG15-1和ISG15-2基因SNP位点分型
根据筛选到的和基因SNP位点, 利用上述两对引物分别对易感组和抗病组大黄鱼个体的和基因序列进行PCR扩增和直接测序。利用DNAStar软件包中的SeqMan软件对测序结果和测序峰图进行分析, 纯合型SNP的测序峰图为单一峰型, 而杂合型SNP的测序峰图为双峰。通过测序峰图分析, 对和基因的SNP位点进行分型。
1.5 SNP位点多态性与哈维氏弧菌抗性的关联性分析
对分型成功的和基因的SNP位点在易感组和抗病组中的基因型频率、等位基因频率等进行统计, 利用SPSS19.0软件进行卡方检验(2test), 对大黄鱼和基因SNP多态性与哈维氏弧菌抗性的关联性开展分析。
1.6 基因单倍型与哈维氏弧菌抗性的关联性分析
利用SHEsis在线分析软件(http://analysis.bio-x. cn/myAnalysis.php)对和基因的SNP位点分别进行连锁不平衡(linkage disequilibrium)分析和单倍型(haplotype)分析。利用卡方检验对和基因的单倍型与哈维氏弧菌抗性的关联性进行分析。
2.1 ISG15-1和ISG15-2基因SNP位点筛选
本研究通过对10尾易感组和10尾抗病组大黄鱼个体的和基因编码序列分别进行PCR扩增, 利用直接测序法检测分析, 在480 bp的基因(图1a, 1b)和468 bp的基因(图1a, 1b)编码序列上分别筛选到10个和4个SNP位点(表2)。在基因的10个SNP位点中, 186 G/C和209 G/T属于碱基颠换(表2), 其余8个SNP位点属于碱基转换(表2)。基因上的4个SNP位点(83 C/T、181 A/G、186 G/C和209 G/T)引起氨基酸发生改变, 属于非同义替代(Nonsynonymous substitution); 其余6个SNP位点未引起氨基酸改变, 属于同义替代(Synonymous substitution) (表2)。在的4个SNP位点中, 1个SNP位点(297 G/T)属于碱基颠换, 其余3个位点均属于碱基转换(表2)。基因的2个SNP位点(83 C/T和170 C/T)属于非同义替代; 而165 C/T和297 G/T位点均属于同义突变(表2)。
经功能结构域定位分析发现, 这些筛选到的SNP位点均位于和蛋白的泛素样结构域(图1c)。在的10个SNP位点中, 7个位点(83 C/T、156 C/T、165 C/T、171 C/T、181 A/G、186 G/C和209 G/T)位于第1个泛素样结构域上, 而3个位点(249 A/G、312 C/T和318 C/T)则位于第2个泛素样结构域上(图1c)。在的4个SNP位点中, 3个位点(83 C/T、165 C/T和170 C/T)位于第1个泛素样结构域上, 而297 G/T位点则位于第2个泛素样结构域上(图1c)。

注: a. 大黄鱼和基因结构图。外显子用黑色方框表示, 内含子用黑色直线表示。NCBI基因组序列的序列号为NC_040013。b. 大黄鱼和蛋白氨基酸序列比对。c. SNP位点在和编码区的分布。泛素样结构域(Ubiquitin-like domain, UBL)用蓝色标记, 连接序列(Linker)用红色标记, 泛素共轭基序(Ubiquitin conjugation motif)用橙色标记

表2 大黄鱼ISG15-1和ISG15-2基因SNP位点信息
注: S表示同义替代, N表示非同义替代
2.2 ISG15-1和ISG15-2基因SNP位点多态性与哈维氏弧菌抗性的关联分析
本研究对易感组和抗病组大黄鱼个体的和基因SNP位点序列进行PCR扩增和直接测序。通过核苷酸比对和测序峰图分析, 成功对的10个SNP位点和的4个SNP位点进行了分型(以基因的181 A/G位点和基因的297 G/T位点的测序峰图为例, 图2)。

图2 大黄鱼ISG15-1和ISG15-2基因测序峰图序列分析
注: a.基因186 G/C位点测序峰图; b.基因297 G/T位点测序峰图。SNP位点用箭头表示
对分型成功的基因10个SNP位点和基因4个SNP位点在100尾易感群体和100尾抗病群体中的基因型频率和等位基因频率进行了统计, 结果如表3和表4所示。利用SPSS19.0软件进行卡方检验发现,基因的186 G/C和318 C/T这2个位点的基因型频率和等位基因频率在易感群体和抗病群体中均存在极显著差异(<0.01, 表3)。基因的181 A/G、209 G/T和312 C/T这3个位点的基因型频率和等位基因频率在易感群体和抗病群体中均存在显著差异(<0.05, 表3)。基因156 C/T和249 A/G位点的等位基因频率在易感群体和抗病群体中差异显著(值分别为0.041和0.031, 表3), 但这2个SNP位点的基因型频率在易感群体和抗病群体中差异均不显著(值分别为0.128和0.073, 表3)。对于基因, 经卡方检验发现, 165 C/T位点的基因型频率和等位基因频率在易感群体和抗病群体中差异显著(值分别为0.021和0.003, 表4), 297 G/T位点的基因型频率和等位基因频率在易感群体和抗病群体中差异极显著(<0.01, 表4)。但是基因的83 C/T和170 C/T这2个位点的基因型频率和等位基因频率在易感群体和抗病群体中的差异均不显著(>0.05, 表4)。

表3 大黄鱼ISG15-1基因SNP位点在易感群体和抗病群体中的频率分布
注: SI表示易感个体(susceptible individuals), RI表示抗病个体(resistant individuals), 数据表示个体数量及其在群体中所占百分比;*表示差异显著(<0.05),**表示差异极显著(<0.01)。下同

表4 大黄鱼ISG15-2基因SNP位点在易感群体和抗病群体中的频率分布
2.3 ISG15-1和ISG15-2基因单倍型与哈维氏弧菌抗性的关联分析
利用SHEsis软件对和基因的SNP位点进行了连锁不平衡分析, 结果如图3所示。的10个SNP位点间均存在不同程度的连锁不平衡(图3a), 形成1个连锁不平衡单倍块(Block 1)和11种单倍型(表5)。其中, 单倍型H3 (CCCCGGTACT)和H5 (CTCCGCGACC)与大黄鱼哈维氏弧菌易感性状极显著相关(<0.01, 表5); 单倍型H2 (CCCCGG TACC)、H6 (TCCCACTGTC)和H9 (TCCCAGTGCC)与大黄鱼哈维氏弧菌抗病性状显著相关(<0.05, 表5)。而的4个SNP位点间同样存在不同程度的连锁不平衡(图3b), 形成1个连锁不平衡单倍块(Block 1)和5种单倍型(表6)。其中, 单倍型H1 (CCCG)和H4 (TCCG)与大黄鱼哈维氏弧菌抗病性状极显著相关(<0.01, 表6); 单倍型H2 (CCCT)与大黄鱼哈维氏弧菌易感性状极显著相关(<0.01, 表6)。

图3 大黄鱼ISG15-1和ISG15-2基因SNP位点连锁不平衡分析
注: a.基因SNP位点连锁不平衡分析, b.基因SNP位点连锁不平衡分析。方块中的数值为连锁不平衡系数(’, 单位: %), 白色(’=0), 粉色(0<’<1), 红色(’=1)

表5 大黄鱼ISG15-1基因单倍型在易感群体和抗性群体中的分布
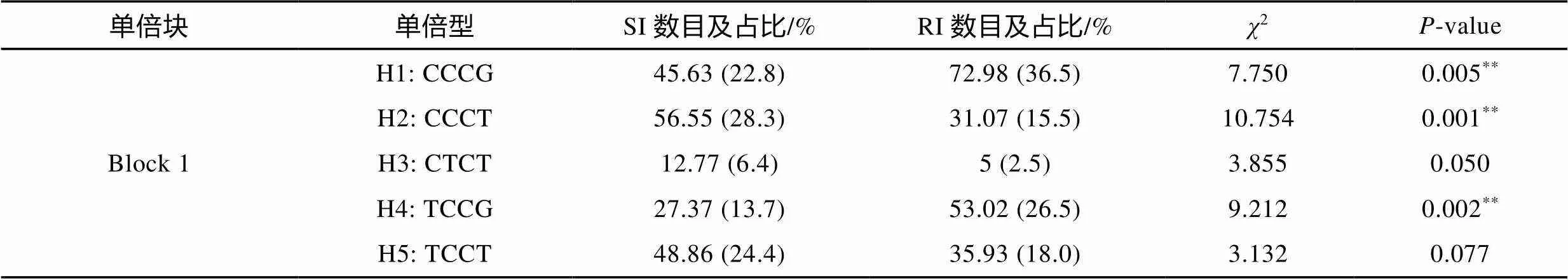
表6 大黄鱼ISG15-2基因单倍型在易感群体和抗性群体中的分布
研究表明干扰素刺激基因在硬骨鱼类抗病毒和细菌天然免疫方面发挥非常重要的作用(Wang, 2012; Huang, 2013)。我们的前期研究发现,和同样参与了大黄鱼抗细菌感染天然免疫反应(Shen, 2019)。然而, 关于大黄鱼基因SNP位点挖掘及与抗病性状关联分析的研究, 目前国内外尚未见报道。本研究从大黄鱼和基因编码序列上分别筛选到10个和4个SNP位点, 分别占和编码序列总碱基的2.08%和0.85%。以上结果表明, 大黄鱼基因(尤其是基因拷贝)的编码序列多态性较低。这可能与基因较高的进化保守性有关, 因为不同硬骨鱼类基因的序列同源相似性非常高(Huang, 2013)。另外, 大黄鱼基因上筛选到的SNP位点数量多于基因, 表明基因编码序列多态性高于基因。这可能与和基因在进化过程中受到的选择压力不同有关。相关生物信息学研究的证据表明,强烈的选择压力会导致基因的遗传多态性降低(Metzger, 2015)。对大黄鱼和基因开展的分子进化分析结果显示,基因拷贝受到了强烈的正选择作用并发生了适应性进化(Shen, 2019)。另外, 有报道指出大多数SNP位点通常位于对蛋白质无直接影响的基因组非编码区域, 而基因编码区域的SNP位点往往比较少(Syvänen, 2001)。对动植物基因组开展的SNPs研究也发现, 非编码区域SNP位点的频率大于编码区域(Ching, 2002; Zhao, 2003)。柴欣等(2017)从团头鲂() MHC IIα基因上总共筛选到35个SNP位点, 仅有13个位于该基因的编码区。陈校辉等(2013)从黄颡鱼() MSTN基因上筛选到8个SNP位点, 其中只有1个位点位于该基因的编码区。孙千惠等(2019)从团头鲂的基因上只筛选到了2个SNP位点, 且均位于非编码区域。因此, 今后可以对大黄鱼和基因的非编码序列开展遗传多态性研究, 以期能筛选到更多的SNP位点。
通过对筛选到的SNP位点进行分型并开展哈维氏弧菌抗性关联分析发现, 大黄鱼基因186 G/C和318 C/T位点的基因型频率以及等位基因频率在易感群体和抗病群体中均存在极显著差异(<0.01)。其中, 186 G/C位点的GG基因型在易感群体中为优势基因型(50.0%), GC基因型在抗病群体中为优势基因型(61.0%); 318 C/T位点的CT基因型在易感群体中为优势基因型(19.0%), CC基因型在抗病群体中为优势基因型(95.0%) (表3)。这些结果表明, 大黄鱼基因186 G/C位点的GC基因型以及318 C/T位点的CC基因型与大黄鱼抗哈维氏弧菌感染呈显著相关性。大黄鱼基因297 G/T位点的基因型频率和等位基因频率在易感群体和抗病群体中均存在极显著差异(<0.01), GT基因型在易感群体中为优势基因型(43.0%), GG基因型在抗病群体中为优势基因型(48.0%) (表4)。该结果表明, 大黄鱼基因297 G/T位点的GG基因型与大黄鱼抗哈维氏弧菌感染呈显著相关性。
对和基因的SNP位点开展连锁不平衡分析发现,的10个SNP位点间以及的4个SNP位点间均存在不同程度的连锁不平衡(图3)。对尼罗罗非鱼()的基因开展SNP筛选及抗性关联分析发现, 从尼罗罗非鱼基因上筛选到的SNP位点间同样存在大量的连锁不平衡(高风英等, 2018)。对吉富尼罗罗非鱼(, GIFT)基因5’调控区序列开展的SNP位点挖掘及抗病性状关联分析表明, 从基因上筛选到的SNP位点间也存在大量的连锁不平衡, 其中2个SNP位点处于完全连锁状态(陈昆平等, 2018)。而对草鱼的TLR22基因开展的遗传多态性研究发现, 草鱼TLR22基因的5个SNP位点间也存在不同程度的连锁不平衡(Su, 2012)。本研究通过连锁不平衡分析, 在大黄鱼中发现了与哈维氏弧菌易感性状极显著相关的单倍型H3 (CCCCGGTACT)和H5 (CTCCGCGACC)以及与抗哈维氏弧菌感染性状显著相关的单倍型H2 (CCCCGGTACC)、H6 (TCCCACTGTC)和H9 (TCCCAGTGCC); 同时在大黄鱼中发现了与抗哈维氏弧菌感染性状极显著相关的单倍型H1 (CCCG)和H4 (TCCG)以及与哈维氏弧菌易感性状极显著相关的单倍型H2 (CCCT)。本研究筛选到的抗病SNP位点(特别是的186 G/C和318 C/T以及的297 G/T)和单倍型(的H2、H6和H9以及的H1和H4)可作为大黄鱼分子育种的候选分子标记, 为抗哈维氏弧菌病大黄鱼品系的遗传选育提供理论基础。
本研究从大黄鱼和基因中分别筛选到10个和4个SNP位点并进行了成功分型。其中,基因的186 G/C和318 C/T位点以及基因的297 G/T位点与大黄鱼哈维氏弧菌抗性呈极显著相关。通过开展连锁不平衡分析发现,基因的10个SNP位点间以及基因的4个SNP位点间均存在不同程度的连锁不平衡。其中,基因的单倍型H2 (CCCCGGTACC)、H6 (TCCCACTGTC)和H9 (TCCCAGTGCC)与大黄鱼哈维氏弧菌抗性显著相关;基因的单倍型H1 (CCCG)和H4 (TCCG)与大黄鱼哈维氏弧菌抗性极显著相关。这些和基因的SNP位点以及单倍型可以作为抗哈维氏弧菌病大黄鱼选育的候选分子标记。
王志勇, 2014. 大黄鱼优质、抗逆品种的培育[J]. 中国科技成果(3): 17-18.
王晨阳, 王璐, 张锐虎, 等, 2019. SNP标记在动物遗传育种及人类疾病动物模型研究中的应用[J]. 中国比较医学杂志, 29(4): 120-125.
孙千惠, 田万平, 罗航, 等, 2019. 团头鲂hepcidin基因单核苷酸多态性及与抗病性状的相关性[J]. 华中农业大学学报, 38(2): 38-44.
孙明洁, 张娜, 徐善良, 等, 2019. 两种弧菌感染大黄鱼免疫相关基因的SNP位点分析[J]. 上海海洋大学学报, 28(5): 772-781.
张玉, 韩明星, 曹明月, 等, 2018. 大黄鱼FST基因SNP筛选及与生长性状相关性分析[J]. 核农学报, 32(5): 883-891.
陈小明, 李佳凯, 王志勇, 等, 2017. 基于简化基因组测序的大黄鱼耐高温性状全基因组关联分析[J]. 水生生物学报, 41(4): 735-740.
陈昆平, 卢迈新, 刘志刚, 等, 2018. 吉富尼罗罗非鱼Ikaros基因5’调控区的克隆、序列分析及抗无乳链球菌相关SNP位点筛选[J]. 中国水产科学, 25(2): 237-250.
陈校辉, 刘朋朋, 王明华, 等, 2013. 黄颡鱼()MSTN基因SNP位点与体重的相关性分析[J]. 海洋与湖沼, 44(6): 1566-1569.
柴欣, 胡晓坤, 马徐发, 等, 2017. 团头鲂Ⅱ基因的SNP位点开发、鉴定及与抗病性状关联分析[J]. 华中农业大学学报, 36(4): 76-82.
徐晓津, 徐斌, 王军, 等, 2010. 大黄鱼感染哈维氏弧菌后血液生化指标的变化及组织病理学观察[J]. 水产学报, 34(4): 618-625.
高风英, 卢迈新, 曹建萌, 等, 2018. 尼罗罗非鱼IPS-1基因SNP位点的筛选及其与链球菌抗性的关联分析[J]. 农业生物技术学报, 26(1): 20-33.
唐嘉嘉, 江飚, 李志成, 等, 2022. 养殖大黄鱼寄生虫病的研究进展[J]. 水产科学, 41(1): 150-159.
嵇祝星, 王晓泉, 刘晓文, 等, 2022. 干扰素刺激基因15(ISG15)在天然免疫中的抗病毒作用研究进展[J]. 中国免疫学杂志, 38(2): 253-258.
BLOMSTROM D C, FAHEY D, KUTNY R,, 1986. Molecular characterization of the interferon-induced 15-kDa protein. Molecular cloning and nucleotide and amino acid sequence [J]. The Journal of Biological Chemistry, 261(19): 8811-8816.
CHING A, CALDWELL K S, JUNG M,, 2002. SNP frequency, haplotype structure and linkage disequilibrium in elite maize inbred lines [J]. BMC Genetics, 3: 19.
DAI Z, LI J C, HU C Y,, 2017. Transcriptome data analysis of grass carp () infected by reovirus provides insights into two immune-related genes [J]. Fish & Shellfish Immunology, 64: 68-77.
D"CUNHA J, KNIGHT JR E, HAAS A L,, 1996. Immunoregulatory properties of ISG15, an interferon-induced cytokine [J]. Proceedings of the National Academy of Sciences of the United States of America, 93(1): 211-215.
HUANG X H, HUANG Y H, CAI J,, 2013. Molecular cloning, expression and functional analysis of ISG15 in orange-spotted grouper,[J]. Fish & Shellfish Immunology, 34(5): 1094-1102.
HUANG X N, WANG Z Y, YAO C L, 2011. Characterization of Toll-like receptor 3 gene in large yellow croaker,[J]. Fish & Shellfish Immunology, 31(1): 98-106.
LIU M, DE MITCHESON Y S, 2008. Profile of a fishery collapse: why mariculture failed to save the large yellow croaker [J]. Fish and Fisheries, 9(3): 219-242.
LIU M J, REIMSCHUESSEL R, HASSEL B A, 2002. Molecular cloning of the fish interferon stimulated gene, 15 kDa (ISG15) orthologue: a ubiquitin-like gene induced by nephrotoxic damage [J]. Gene, 298(2): 129-139.
METZGER J, KARWATH M, TONDA R,, 2015. Runs of homozygosity reveal signatures of positive selection for reproduction traits in breed and non-breed horses [J]. BMC Genomics, 16: 764.
O"FARRELL C, VAGHEFI N, CANTONNET M,, 2002. Survey of transcript expression in rainbow trout leukocytes reveals a major contribution of interferon-responsive genes in the early response to a rhabdovirus infection [J]. Journal of Virology, 76(16): 8040-8049.
SCHNEIDER W M, CHEVILLOTTE M D, RICE C M, 2014. Interferon-stimulated genes: a complex web of host defenses [J]. Annual Review of Immunology, 32: 513-545.
SHEN B, WEI K, GUO S Y,, 2019. Molecular characterization and expression analyses of two homologues of interferon- stimulated gene ISG15 in(Family: Sciaenidae) [J]. Fish & Shellfish Immunology, 86: 846-857.
SU J G, HENG J F, HUANG T,, 2012. Identification, mRNA expression and genomic structure ofand its association with GCRV susceptibility/resistance in grass carp () [J]. Developmental & Comparative Immunology, 36(2): 450-462.
SYVÄNEN A C, 2001. Accessing genetic variation: genotyping single nucleotide polymorphisms [J]. Nature Reviews Genetics, 2(12): 930-942.
WANG K J, CAI J J, CAI L,, 2009. Cloning and expression of a hepcidin gene from a marine fish () and the antimicrobial activity of its synthetic peptide [J]. Peptides, 30(4): 638-646.
WANG W, ZHANG M, XIAO Z Z,, 2012.ISG15: a secreted cytokine-like protein that stimulates antiviral immune response in a LRGG motif- dependent manner [J]. PLoS One, 7(9): e44884.
ZHANG D X, ZHANG D E, 2011. Interferon-stimulated gene 15 and the protein ISGylation system [J]. Journal of Interferon & Cytokine Research, 31(1): 119-130.
ZHAO Z M, FU Y X, HEWETT-EMMETT D,, 2003. Investigating single nucleotide polymorphism (SNP) density in the human genome and its implications for molecular evolution [J]. Gene, 312: 207-213.
ZHENG W B, TIAN C, CHEN X H, 2007. Molecular characterization of goose-type lysozyme homologue of large yellow croaker and its involvement in immune response induced by trivalent bacterial vaccine as an acute-phase protein [J]. Immunology Letters, 113(2): 107-116.
SINGLE NUCLEOTIDE POLYMORPHISMS AND ITS ASSOCIATION WITH DISEASE RESISTANT TRAIT OFGENES IN
LI Feng-Xin1, YIN Xiao-Long2, LU De-Zheng1, LIU Cheng1, ZHANG Jian-She1, SHEN Bin1
(1. National Engineering Research Center of Marine Facilities Aquaculture, Zhejiang Ocean University, Zhoushan 316022, China; 2. Zhoushan Fisheries Research Institute of Zhejiang Province, Zhoushan 316111, China)
Individuals of large yellow croaker () were infected by bacterial, from which susceptible and resistant ones were grouped. The single nucleotide polymorphisms (SNPs) of interferon-stimulated genecopies (and) in large yellow croaker were screened and genotyped by PCR amplification and direct DNA sequencing, and the association of SNPs with the resistance of large yellow croaker towas analyzed. Results show that, 10 and 4 SNPs were screed and successfully genotyped from theandgene, respectively. The statistical analyses showed that the differences of genotype and allele frequencies of 186 G/C and 318 C/T ingene and 297 G/T ingene were extremely significant between the susceptible and resistant groups, indicating that the three SNPs were significantly associated with the resistance of large yellow croaker toinfection. In addition, the linkage disequilibrium analyses showed that the 10 SNPs ofgene formed 1 haplotype block and 11 haplotypes, and the 4 SNPs ofgene formed 1 haplotype and 5 haplotypes. Thereinto, haplotypes of H2 (CCCCGGTACC), H6 (TCCCACTGTC), and H9 (TCCCAGTGCC) ofgene, and haplotypes of H1 (CCCG) and H4 (TCCG) ofgene were all significantly associated with the resistance of large yellow croaker toinfection. These SNPs and haplotypes ofandgenes could be used as candidate molecular markers for selective breeding of disease resistant varieties of large yellow croaker.Key words; interferon-stimulated gene; single nucleotide polymorphism;
S917.4
10.11693/hyhz20220400104
*浙江省自然科学基金项目, LY19D060004号。李凤欣, 硕士研究生, E-mail: 17865815170@163.com
沈 斌, 硕士生导师, E-mail: shenbin@zjou.edu.cn
2022-04-22,
2022-06-18
猜你喜欢大黄鱼弧菌抗病销量增长200倍!“弧菌克星”风靡行业,3天杀灭98%弧菌当代水产(2022年8期)2022-09-20我国小麦基因组编辑抗病育种取得突破今日农业(2022年4期)2022-06-01副溶血弧菌检测方法的研究进展食品安全导刊(2021年20期)2021-08-3028元/斤的辉煌不再!如今大黄鱼深陷价格“泥沼”,休渔期或初现曙光当代水产(2019年6期)2019-07-25如何有效防控对虾养殖中的弧菌病当代水产(2019年5期)2019-07-25宁德迎来大黄鱼丰收季农产品市场周刊(2017年45期)2017-12-28bZIP转录因子在植物激素介导的抗病抗逆途径中的作用浙江农业学报(2017年1期)2017-05-17葡萄新品种 优质又抗病新农业(2016年20期)2016-08-16膳食锌对饲料引起的大黄鱼铜中毒症的保护作用饲料博览(2016年5期)2016-04-05副溶血弧菌噬菌体微胶囊的制备及在饵料中的应用广东海洋大学学报(2015年3期)2015-12-22